CNAS+国家CMA二合一扩项现场评审
2022/07/06

疫情以来,mRNA疫苗迅速破圈走红,刮起了一场“催生新一代疗法的医学革命”。[1]实际上,早在1960年代,科研人员就首次发现了mRNA。此后历经半个多世纪的探索,直至2020年新冠mRNA疫苗上市,这款被寄予厚望的明星产品终于走向商业化落地的初舞台。[2]

mRNA技术大放异彩的背后,是工艺技术的大跨步进展。研究表明,核酸的Poly A结构可以让mRNA在细胞中免受核酶的降解,增强mRNA的稳定性,同时Poly A结构长度可以控制转录效率。因此,有效地检测核酸尾部Poly A的长度及不同长度A的占比是评估mRNA质量的重要指标。
随着越来越多mRNA产品的研发申报,客户对于polyA分析方法的可靠性和完成周期提出了更高的要求,这也对研究型实验室提出了更高的挑战。本期#微谱药研院·技术共探,我们将共同探讨LC-MS法检测mRNA核酸Poly A尾长的原理,以两则案例展开分析,并就常见问题给予答疑。如果还有更多疑问,欢迎文末留言讨论!
1. 方法原理

mRNA样本的Poly A酶切处理一般采用Rnase T1酶。T1酶的特异性酶切位点是G碱基右侧的磷酸二酯键,而酶切后的Poly A尾片段回收方式一般有两种:一是直接将Poly A尾片段从mRNA中进行直接分离后上机分析;二是在前一步基础上,再利用Oligo磁珠提取Poly A尾巴片段进行上机分析(LC-MS)。
2. 案例分析
CASE 01:T1酶直接酶切,无纯化
T1酶直接酶切,前处理简单,但是杂质较多。对于碱基个数多的核酸片段,出峰时间会靠后,见图1。对单个峰进行原始质荷比图的提取,通过原始质荷比图的特征定位Poly A尾巴片段。
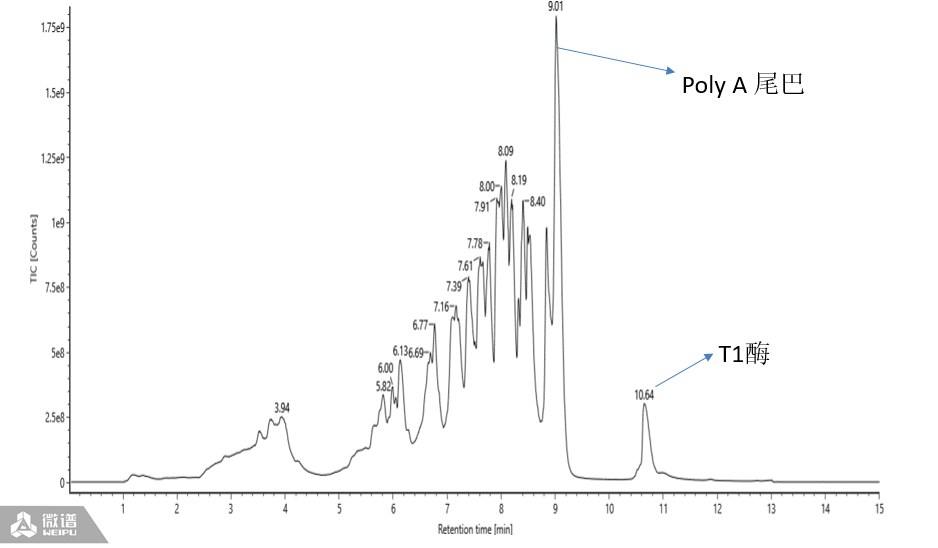
图1:直接酶切后的TIC图
Poly A尾巴片段的原始质荷比图为正态分布或者类正态分布,见图2。
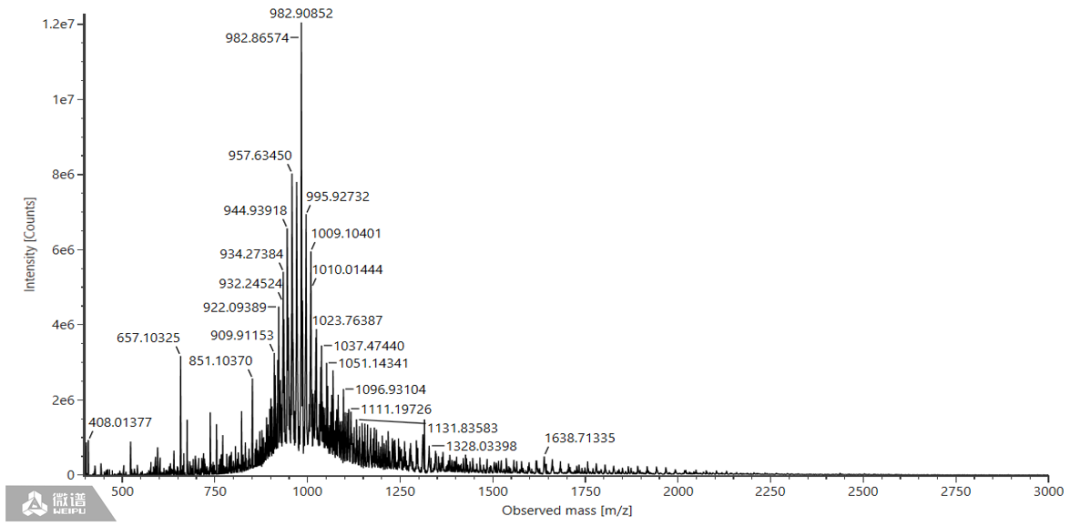
图2:原始质荷比图
再对质谱数据进行解卷积分析可得到结果,见图3。
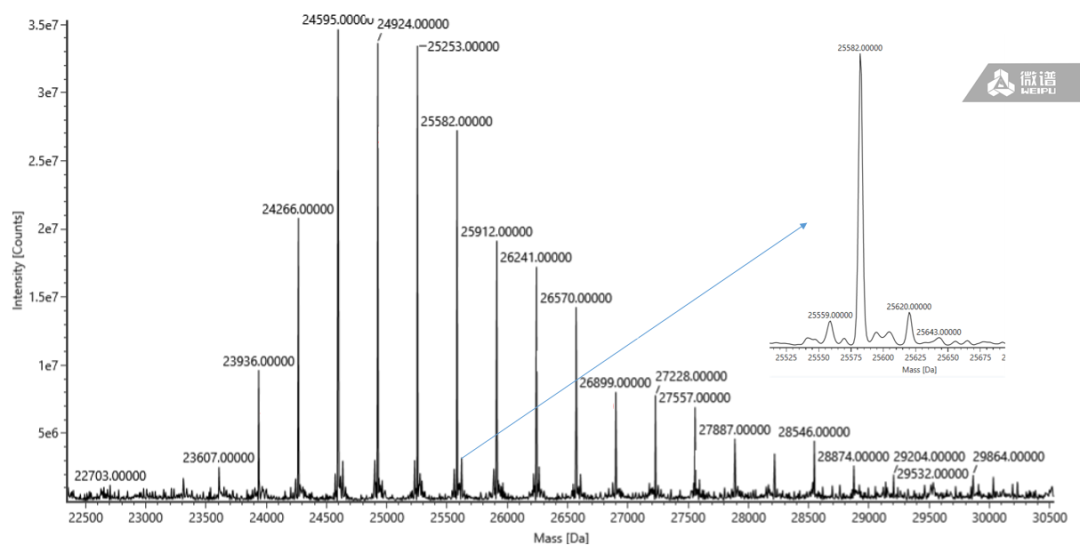
图3:解卷积图谱
CASE 02:T1酶切和Oligo磁珠纯化
与前面的方法相比,加入olig磁珠虽然使得前处理变复杂,成本也会相对升高,但最终呈现的效果会有明显提升,杂峰也显著减少,见图4。
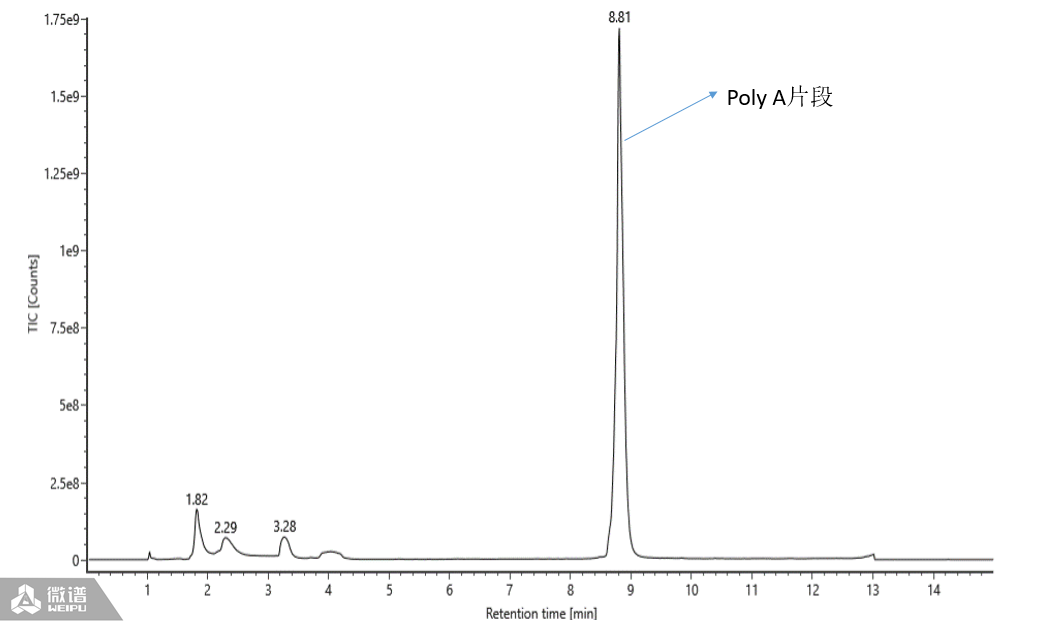
图4:酶切加磁珠纯化后的TIC图
对于一个mRNA样品, 假设理论序列如图5所示。采用T1酶酶切,可能会有2段Poly A尾巴片段。但是多段尾巴可能在TIC图上是重叠的,原始质荷比图也会有所重叠,但分子量不会重叠,如图4所示,RT为8.81min的峰,其实是两段Poly A尾巴片段(碱基差异值是35A)的重叠。

图5:某个核酸的理论序列与酶切位点
1) 当碱基个数相差更多时,可在TIC图上看到区别,同样原始质荷比图可以看到有所叠加,如图6所示。根据碱基个数多的核酸片段,出峰时间会靠后的特点,合理选择提取原始质荷比图的范围或者优化液相参数,可对被T1酶切成两段的Poly A尾巴片段进行数据分析。
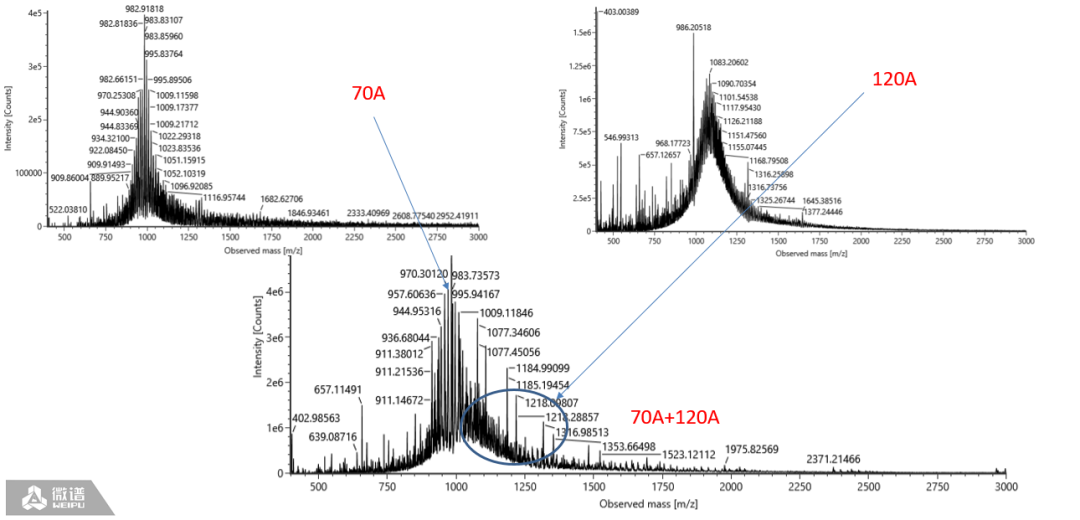
图6:两段不同Poly A尾巴片段的原始质荷比图(单个和叠图)
2) 也可优化液相参数将两段加尾片段在UV和TIC图谱上进行分离,如图7所示。两段加尾片段的原始质荷比图和解卷积图谱见图8和图9。
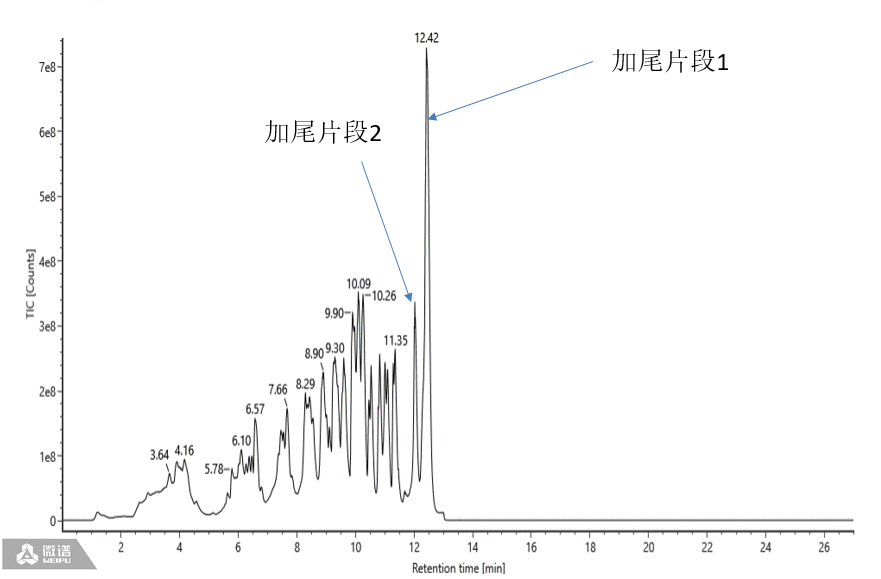
图7两段加尾片段的TIC图谱
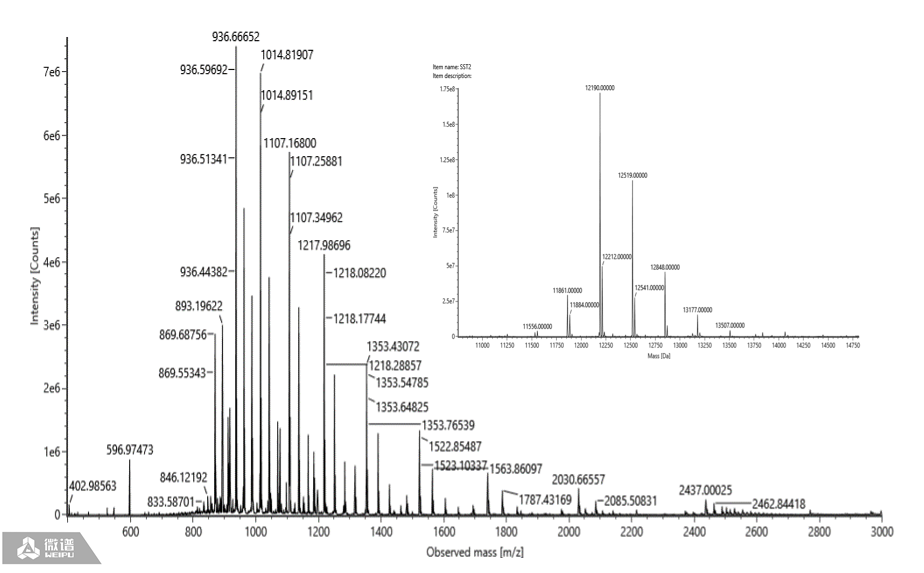
图8加尾片段1的原始质荷比图和解卷积图谱
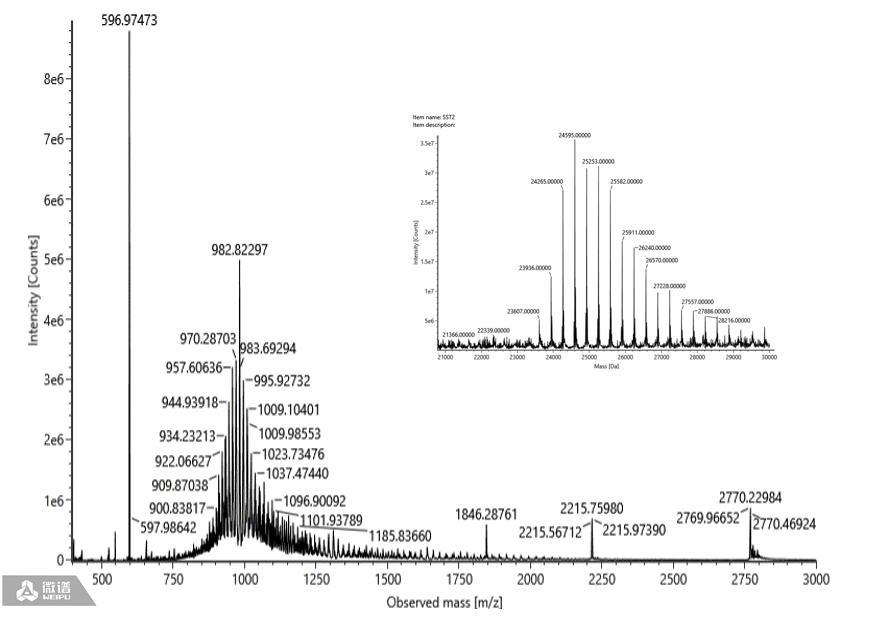
图9加尾片段2的原始质荷比图和解卷积图谱
Poly A尾巴片段主要报告不同长度尾巴片段的占比,根据不同长度A在解卷积图谱上的响应值来计算。一般理论分子量和实际分子量的差值不超出2Da。
计算公式如下所示:
比例(%)=(X/S)× 100%
X:不同Poly A长度的峰高,不同的Poly A长度利用其分子量来区分。
S:所有检测到的不同Poly A长度的峰高之和。